L’apparition des nouveaux sous-variants d’Omicron, XBB et BQ.1.1, et la possible infection simultanée par deux variants, conjuguées à une co-infection avec la grippe complique la tâche des chercheurs. « Quelle sera la situation sanitaire cet hiver ? » s’inquiètent deux chercheurs qui soulignent qu’en l’absence de financements publics et d’un conseil scientifique, il va être difficile de prévoir l’évolution du coronavirus et d’anticiper de nouvelles vagues de contamination.
Une certaine « fatigue pandémique » a gagné une partie de la population, mais le SARS-CoV-2, lui, continue d’évoluer. Alors que la France connaît sa huitième vague (la quatrième de 2022), dominée par le sous-variant Omicron BA.5, elle voit progresser rapidement un autre sous-variant, nommé BQ.1.1.
Samuel Alizon, directeur de recherche (CNRS, CIRB) et Mircea Sofonea, maître de conférences (Université de Montpellier, MIVEGEC), reviennent sur la situation sanitaire qui s’annonce cet hiver et pointent les enjeux de la surveillance et de la recherche dans notre pays. Pour quelles conséquences ?
The Conversation : Avec la fin de l’été, l’actualité a refait place au Covid et à ses variants. C’est désormais BQ.1.1 et autres XBB qui sont évoqués. Que peut-on déjà dire de ces « sous-variants » ? Et comment sont-ils suivis ?
Samuel Alizon : Depuis septembre, on assiste à une forte diversification du SARS-CoV-2, avec l’émergence de nombreux sous-variants du variant Omicron.
BA.4.6, BA.2.75, BA.5.2 et même B.1.1.529.5.3.1.1.1.1.1.1, rebaptisé BQ.1.1 selon la nomenclature Pango, qui propose un système d’identification pour suivre les lignées génétiques du SARS-CoV-2 présentant un intérêt épidémiologique…
Toutes ces lignées, qui prédominent dans diverses régions du monde (BQ.1.1, par exemple, est en pleine progression actuellement en France) appartiennent officiellement au variant Omicron. Il s’agit donc de sous-variants, mais en réalité, on pourrait facilement les qualifier de variants.
The #Omicron evolutionary tree is expanding rapidly, and all 5 of the Omicron greatgrandchildren are capable of evading much of the immune response against #SARSCoV2 and — sadly — some of the protection provided by prior #COVID19 illness or early forms of #vaccines . pic.twitter.com/cSXElpE3FU
— Laurie Garrett (@Laurie_Garrett) October 12, 2022
SA : À ce stade, bien des connaissances sur ces nouvelles lignées sont à prendre avec précaution car elles sont, au mieux, issues de pré-publications, non relues par les pairs. On ne sait que peu de chose de leur virulence et, évidemment, quasiment rien sur les effets à long terme engendrés par leurs infections.
4 facteurs qui favorisent le Covid long ont été identifiés
Nous sommes certains, en revanche, des mutations qui sont présentes dans les génomes de ces lignées, puisque ce sont elles qui les définissent. Le variant BA.5 avait par exemple fixé une mutation en position 452 de la protéine Spike. Celle-ci était très étudiée, car elle était caractéristique du variant Delta au moment de son apparition.
Pour BQ.1.1, on observe toute une autre série de mutations dans le Receptor Binding Domain (RBD) de cette protéine, autrement dit la partie de la protéine Spike qui interagit avec ACE2, la « serrure » située à la surface des cellules qu’infecte le SARS-CoV-2. C’est notamment le cas de la mutation S :R346T. Comme l’indique une pré-publication issue d’un consortium d’équipes chinoises, ces mutations semblent conférer un important potentiel d’évasion immunitaire à cette lignée. De plus, BQ.1.1 pourrait ne pas être sensible aux traitements par les anticorps monoclonaux disponibles en France (telle la combinaison tixagevimab-cilgavimab (Evusheld)).
Parmi les sous-variants surveillés en raison notamment de ses potentielles capacités d’échappement immunitaire, on peut aussi mentionner la lignée XBB, qui est issue d’une recombinaison entre virus des lignées BJ.1 et BM.1.1 lors de la co-infection d’une même cellule.
TC : Comment surveille-t-on ces sous-variants préoccupants ? D’où proviennent les données épidémiologiques ?
SA : Au niveau épidémiologique, la qualité du système de surveillance britannique est toujours remarquable. Leur dernier rapport du 7 octobre 2022, qui combine données de dépistage et de séquençage, offre une vision particulièrement claire de leur situation épidémique.
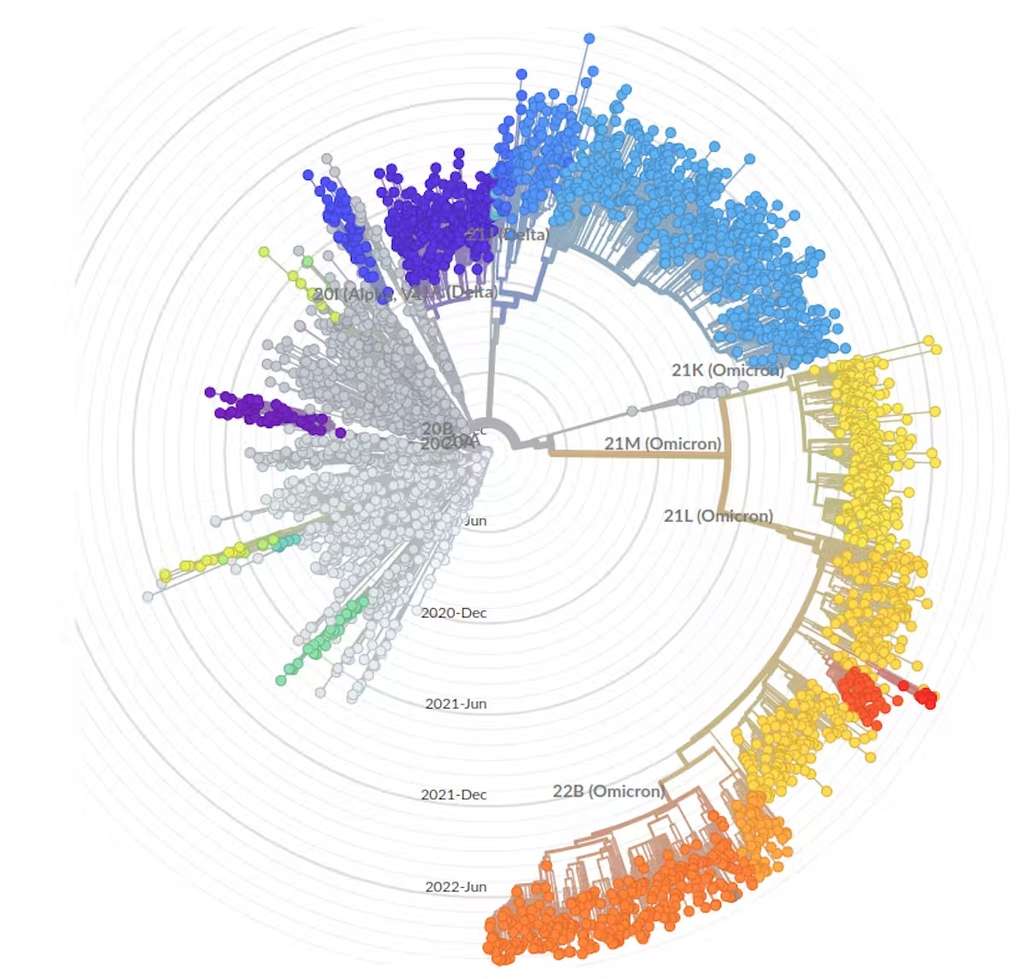
PHYLOGÉNIE RADIALE DE NEXTSTRAIN.ORG À PARTIR DES DONNÉES GISAID QUI MONTRE QUE LA DIVERSIFICATION DES LIGNÉES OMICRON EST BIEN SUPÉRIEURE À CELLE DU VARIANT DELTA (EN BLEU) OU ALPHA (EN VIOLET).© NEXTSTRAIN.ORG/NCOV
Pour les autres pays, dont la France, on s’en remet aux données de séquençage partagées sur la plate-forme GISAID. Plusieurs sites Internet, dont Nextstrain.org mais aussi l’excellent covSPECTRUM de l’équipe du Pr Tanja Stadler, en Suisse, permettent de visualiser la dynamique des variants en temps réel (dans la limite des données fournies par chaque pays).
Mircea Sofonea : Notons que contrairement aux précédentes vagues causées par l’arrivée d’un nouveau variant, les données de criblage issues des dépistages RT-qPCR (qui repèrent des mutations définies au préalable, et servent donc à traquer des variants déjà connus) ne permettent plus de distinguer ces nouvelles lignées.
Cela nous prive d’un signal précoce et donc précieux pour informer en temps réel les modèles de la dynamique courante de remplacement. Celle-ci ne peut alors être connue qu’au moyen du séquençage, avec un retard d’au moins une semaine après le prélèvement (lui-même arrivant plusieurs jours après le début de l’infection) et sur un échantillon qui, pour des raisons matérielles, est réduit – les enquêtes Flash réalisées par le consortium Emergen portant sur 1 000 à 2 000 séquences interprétables.
Le problème est que, cette fois-ci, la France se retrouve la première à connaître la prédominance du nouveau (sous-)variant (BQ.1.1). On ne pourra donc plus compter sur les tendances observées outre-Manche !
Alors que la diversité génétique du SARS-CoV-2 met une nouvelle fois à l’épreuve notre système de surveillance et de soins, la chaîne technico-scientifique, du prélèvement individuelle à l’analyse populationnelle, sur laquelle repose notre anticipation collective, a besoin d’un investissement immédiat à la hauteur de l’enjeu de santé publique.
TC : Comment ces variants s’inscrivent dans l’évolution générale observée pour Omicron ?
SA : Jusque-là, on avait souvent une lignée dominante et des remplacements de variants assez prononcés : Alpha a remplacé toutes les lignées qui le précédaient, puis Delta a remplacé Alpha et ainsi de suite.
Là, comme on peut le visualiser sur le site Nexstrain.org, les sous-lignées issues de BA.2, BA.4 et BA.5 semblent co-circuler au niveau mondial.
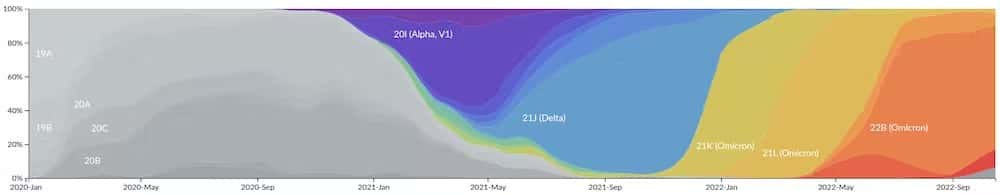
FRÉQUENCE DES PRINCIPAUX CLADES SELON LE SITE NEXTSTRAIN.ORG À PARTIR DES DONNÉES GISAID.
Concernant les raisons de cette diversification, impossible évidemment d’avoir des certitudes. L’étude de la diversité biologique est un champ à part entière mais on peut émettre deux hypothèses.
D’une part, la diversité des parasites est souvent corrélée à celle de leurs hôtes. Or, les populations humaines sont maintenant plus diversifiées que jamais au niveau de leur immunité, qu’elle soit d’origine vaccinale ou post-infectieuse.
D’autre part, la diversification du virus est aussi proportionnelle au nombre de nouvelles infections et actuellement, dans beaucoup de pays, le virus circule de manière incontrôlée.
MS : S’il est impossible à ce jour, et certainement pour encore de nombreuses années, de prévoir la trajectoire évolutive précise d’un coronavirus, la dynamique de diversification observée depuis le printemps n’est pas forcément étonnante.
Les deux premières années de la pandémie ont été marquées par de fortes pressions de sélections portées sur la transmission – Alpha et Delta n’ont pu émerger que parce que leur contagiosité particulièrement élevée leur permettait de compenser la distanciation sociale et les masques.
Omicron, qui est issu d’une vieille branche de l’arbre phylogénétique du SARS-CoV-2 et non de ces deux variants, s’est quant à lui propagé dans un contexte où l’immunité populationnelle (post-infectieuse et vaccinale) était devenue un frein supplémentaire à la propagation virale. Depuis le printemps 2022, cette immunité est d’ailleurs le seul frein restant.
Grâce à la combinaison de sa contagiosité élevée et de son fort échappement immunitaire, Omicron BA.1 a quasiment poussé à l’extinction les autres lignées et bénéficié d’une circulation sans guère de contrainte outre de l’immunité humorale et cellulaire développée contre eux.
Or, comme chaque infection est source de mutations (aléatoires), les conditions étaient réunies pour mettre en place un « tapis-roulant » de diversification alimenté par l’échappement immunitaire.
TC : Cette capacité de diversification est-elle donc sans fin ?
SA : Cette question invite à revenir sur les débats de 2020 voire 2021 où d’aucuns annonçait un « essouflement » du virus dont les possibilités mutationnelles iraient en diminuant.
En fait, l’évolution virale est toujours difficile à anticiper car chaque mutation peut complètement modifier le « paysage adaptatif » du virus, c’est-à-dire ses possibilités d’évolution. Aux contraintes génétiques se rajoutent celles de l’environnement et de la variabilité des populations infectées par le virus.
Un point intéressant dans la diversification actuelle des lignées virales est qu’il existe un certain nombre de cas d’évolution parallèle, c’est-à-dire de lignées qui fixent indépendamment la même mutation.
Convergent evolution UPDATE 10/19. More converging BQ.1.* sublineages and an interesting pipeline (CA.4, BN.4, BN.3.1, BL.1.*) @EricTopol @TRyanGregory @DrEricDing @ewencallaway @CorneliusRoemer @siamosolocani @florian_krammer @kakape @denise_dewald @mildanalyst @RobertoBurioni pic.twitter.com/r3ymEOEyUp
— Daniele Focosi, MD PhD MSc (@dfocosi) October 19, 2022
SA : C’est très difficile, car au-delà de la difficulté scientifique, les équipes de recherche en France n’ont quasiment plus de financements annuels de base (appelés « récurrents »), et de nombreux projets ont été refusés cette année. Bref, nous ne sommes plus en mesure d’explorer des scénarios prospectifs. Et contrairement à 2020 et 2021, il n’y a plus de conseil scientifique pour solliciter de telles analyses. Les inconnues sur le déroulement de l’hiver sont donc énormes.
Ce qu’on peut dire malgré tout, c’est que la vague actuelle (qui a débuté mi-septembre) est encore dominée par la lignée BA.5, qui avait déjà causé le troisième pic épidémique de 2022, en juillet. Cette quatrième vague a probablement des origines multiples, mais on peut noter qu’elle a coïncidé avec la rentrée scolaire et une circulation observée d’abord chez les enfants.
Quant à savoir pourquoi le même variant a pu générer une nouvelle vague si peu de temps après la précédente, il est probable que les facteurs sociaux ont été déterminants. L’été ayant probablement cassé la vague précédente et la rentrée favorisé la reprise de la circulation virale. La persistance d’une immunité post-infection issue de la vague estivale pour BA.5 explique probablement pourquoi le pic a été atteint si rapidement pour les enfants (avant fin septembre).
Pour les adultes, l’épidémie a, logiquement, démarré plus tardivement et par un débordement de l’épidémie chez les enfants. Malheureusement, le pic épidémique a cette fois tardé à se dessiner. Les données issues du séquençage sont encore parcellaires mais, comme le suggère les visualisations du site covSPECTRUM, il se pourrait que la montée des « nouveaux » variants mentionnés ci-dessus (tel BQ.1.1) en soit la cause.
futura